cd /home/iu/chipseq/data
ls data
mkdir fastqc_before
fastqc --nogroup -t 2 -o ./fastqc_before \
data/input_1.fastq.gz \
data/input_2.fastq.gz \
data/sample_1.fastq.gz \
data/sample_2.fastq.gz
firefox \
fastqc_before/input_1_fastqc/fastqc_report.html \
fastqc_before/input_2_fastqc/fastqc_report.html \
fastqc_before/sample_1_fastqc/fastqc_report.html \
fastqc_before/sample_2_fastqc/fastqc_report.html
mkdir trimmed_data
java -jar /usr/local/bin/trimmomatic-0.36.jar PE \
-threads 2 -phred33 \
data/input_1.fastq.gz \
data/input_2.fastq.gz \
trimmed_data/input_1_paired.fastq \
trimmed_data/input_1_unpaired.fastq \
trimmed_data/input_2_paired.fastq \
trimmed_data/input_2_unpaired.fastq \
LEADING:20 TRAILING:20 SLIDINGWINDOW:4:15 MINLEN:36
java -jar /usr/local/bin/trimmomatic-0.36.jar PE \
-threads 2 -phred33 \
data/sample_1.fastq.gz \
data/sample_2.fastq.gz \
trimmed_data/sample_1_paired.fastq \
trimmed_data/sample_1_unpaired.fastq \
trimmed_data/sample_2_paired.fastq \
trimmed_data/sample_2_unpaired.fastq \
LEADING:20 TRAILING:20 SLIDINGWINDOW:4:15 MINLEN:36
cat /proc/cpuinfo
mkdir fastqc_after
fastqc --nogroup -t 2 -o fastqc_after \
trimmed_data/input_1_paired.fastq \
trimmed_data/input_2_paired.fastq \
trimmed_data/sample_1_paired.fastq \
trimmed_data/sample_2_paired.fastq
firefox \
fastqc_after/input_1_paired_fastqc/fastqc_report.html \
fastqc_after/input_2_paired_fastqc/fastqc_report.html \
fastqc_after/sample_1_paired_fastqc/fastqc_report.html \
fastqc_after/sample_2_paired_fastqc/fastqc_report.html
mkdir mapping
bowtie2 -p 2 -x /home/iu/genome/sacCer3/Bowtie2Index/genome \
-1 trimmed_data/input_1_paired.fastq \
-2 trimmed_data/input_2_paired.fastq | \
samtools view -Sb - > mapping/input.bam
samtools sort mapping/input.bam -o mapping/input.sorted.bam
bowtie2 -p 2 -x /home/iu/genome/sacCer3/Bowtie2Index/genome \
-1 trimmed_data/sample_1_paired.fastq \
-2 trimmed_data/sample_2_paired.fastq | \
samtools view -Sb - > mapping/sample.bam
samtools sort mapping/sample.bam -o mapping/sample.sorted.bam
macs2 callpeak \
-t mapping/sample.sorted.bam \
-c mapping/input.sorted.bam \
--outdir macs2_res \
-f BAMPE \
-n handson2016 \
-B \
-q 0.01 \
-g 1.2e+7
head -5 handson2016_peaks.narrowPeak
cd mapping
samtools index input.sorted.bam
samtools index sample.sorted.bam
cd peak_detection
cat handson2016_summits.bed | sort -k 5n
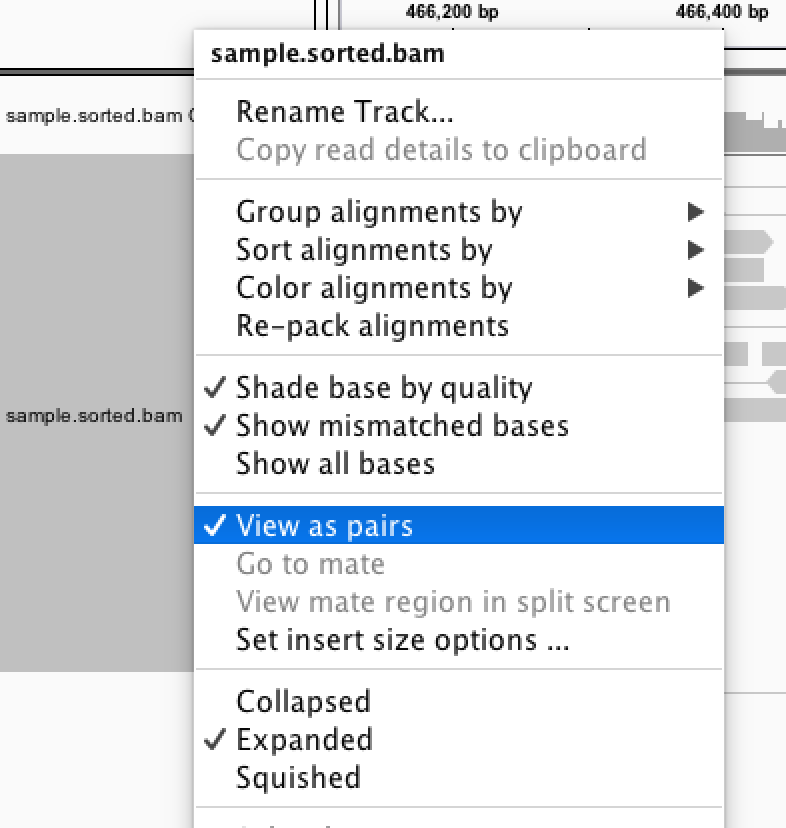
IGVでリードのペアを繋いで表示するには、 view as pair を有効にします
mkdir annotation
cd annotation
cat ../macs2_res/handson2016_summits.bed
java -jar /usr/local/bin/snpEff.jar eff \
-csvStats stats.txt \
-c /usr/local/bin/snpEff.config \
-i bed \
-o bedAnn R64-1-1.82 \
../macs2_res/handson2016_summits.bed > \
handson2016_summits.annotated.bed
less handson2016_summits.annotated.bed
mkdir ../motif
cd ../motif
R
library(rGADEM)
library("BSgenome.Scerevisiae.UCSC.sacCer3")
BED <- read.table("../macs2_res/handson2016_peaks.narrowPeak", header=FALSE, sep="\t")
BED <- data.frame(chr=as.factor(BED[,1]),start=as.numeric(BED[,2]),end=as.numeric(BED[,3]))
rgBED<-IRanges(start=BED[,2],end=BED[,3])
Sequences<-RangedData(rgBED,space=BED[,1])
gadem<-GADEM(Sequences,verbose=1,genome=Scerevisiae)
pdf("motif.pdf")
plot(gadem)
dev.off()
q()evince motif.pdf
このセクションは2016年7月28日の講義中に受講生の熱い要望を受け追加しました。
講義中で使用した snpEff 以外のアノテーション方法を示したものです。
また、この内容は講義資料のPDFには含まれておりませんのでご注意ください。
下記のコマンドを /home/iu/chipseq/ で実行します。
mkdir chippeakanno
cd chippeakanno
R
Rを起動して下記のコマンドを実行します。
/home/iu/chipseq/chippeakanno に下記のファイルが出力されます。
annotated.bed- アノテーション済みのBEDファイル
annotation_summary.txt- アノテーションされた領域の集計情報
annotation.pdf- アノテーションされた領域を図示したもの
library(ChIPpeakAnno)
ensembl = useMart(biomart="ENSEMBL_MART_ENSEMBL", host="www.ensembl.org")
sacCer3 <- useDataset("scerevisiae_gene_ensembl", mart=ensembl)
ann.data <- getAnnotation(mart=sacCer3, featureType="TSS")
bed.df <- read.table("../macs2_res/handson2016_peaks.narrowPeak", header=FALSE, sep="\t")
bed.gr <- BED2RangedData(bed.df[1:4], header = FALSE)
bed.anno <- annotatePeakInBatch(RangedData(bed.gr), AnnotationData = ann.data, output = "both")
bed.anno.table <- table(as.data.frame(bed.anno)$insideFeature)
write.table(bed.anno.table, "annotation_summary.txt", quote=FALSE, sep="\t")
write.table(as.data.frame(bed.anno), "annotated.bed", quote=FALSE, sep="\t", row.names=FALSE)
library(ggplot2)
fortify.table <- function(model, ...) {
data <- reshape2::melt(model)
return(data)
}
reshape.bed <- reshape2::melt(bed.anno.table)
reshape.bed
pdf("annotation.pdf")
g <- ggplot(data=reshape.bed, aes(x=Var1, y=value, fill=Var1))
g <- g + guides(fill=FALSE)
g <- g + geom_bar(width=0.8, stat='identity')
g <- g + xlab('Region')
g <- g + ylab('Peak count')
g <- g + ggtitle('Peak Binding Regions')
g <- g + theme(axis.text.x = element_text(angle=60, hjust=1))
plot(g)
dev.off()出力されるグラフの例
おおまかな流れとともにコマンドを紹介します。
ライブラリを読み込みます。
今回はデータの取得に biomaRt を使用していますが、 ChIPpeakAnno の読み込み時に同時にロードされるので個別の読み込みは不要です。
library(ChIPpeakAnno)アノテーションデータを取得します。
ChIPpeakAnno には幾つかの生物種についてデータが同梱されていますが、sacCer3のデータは存在しないため、別途取得する必要があります。
ここでは、 biomaRt を使用して、ensemblよりTSSのデータを取得します。
ensembl = useMart(biomart="ENSEMBL_MART_ENSEMBL", host="www.ensembl.org")
sacCer3 <- useDataset("scerevisiae_gene_ensembl", mart=ensembl)
ann.data <- getAnnotation(mart=sacCer3, featureType="TSS")ピークデータを読み込みます。
bed.df <- read.table("../macs2_res/handson2016_peaks.narrowPeak", header=FALSE, sep="\t")領域の情報を計算します。
不要なピークのスコアなどは使用しないように bed.df[1:4] と指定します。
bed.gr <- BED2RangedData(bed.df[1:4], header = FALSE)アノテーションを実施します。
bed.anno <- annotatePeakInBatch(RangedData(bed.gr), AnnotationData = ann.data, output = "both")ゲノム上の領域情報のみをサマリーとして取得します。
bed.anno.table <- table(as.data.frame(bed.anno)$insideFeature)それぞれのデータをファイルに書き出します。
write.table(bed.anno.table, "annotation_summary.txt", quote=FALSE, sep="\t")
write.table(as.data.frame(bed.anno), "annotated.bed", quote=FALSE, sep="\t", row.names=FALSE)最後に ggplot2 を使用してグラフを作成します。
library(ggplot2)
fortify.table <- function(model, ...) {
data <- reshape2::melt(model)
return(data)
}
reshape.bed <- reshape2::melt(bed.anno.table)
reshape.bed
pdf("annotation.pdf")
g <- ggplot(data=reshape.bed, aes(x=Var1, y=value, fill=Var1))
g <- g + guides(fill=FALSE)
g <- g + geom_bar(width=0.8, stat='identity')
g <- g + xlab('Region')
g <- g + ylab('Peak count')
g <- g + ggtitle('Peak Binding Regions')
g <- g + theme(axis.text.x = element_text(angle=60, hjust=1))
plot(g)
dev.off()